Methanol Generates Numerous Artifacts during Sample Extraction and Storage of Extracts in Metabolomics Research
Abstract
1. Introduction
2. Results
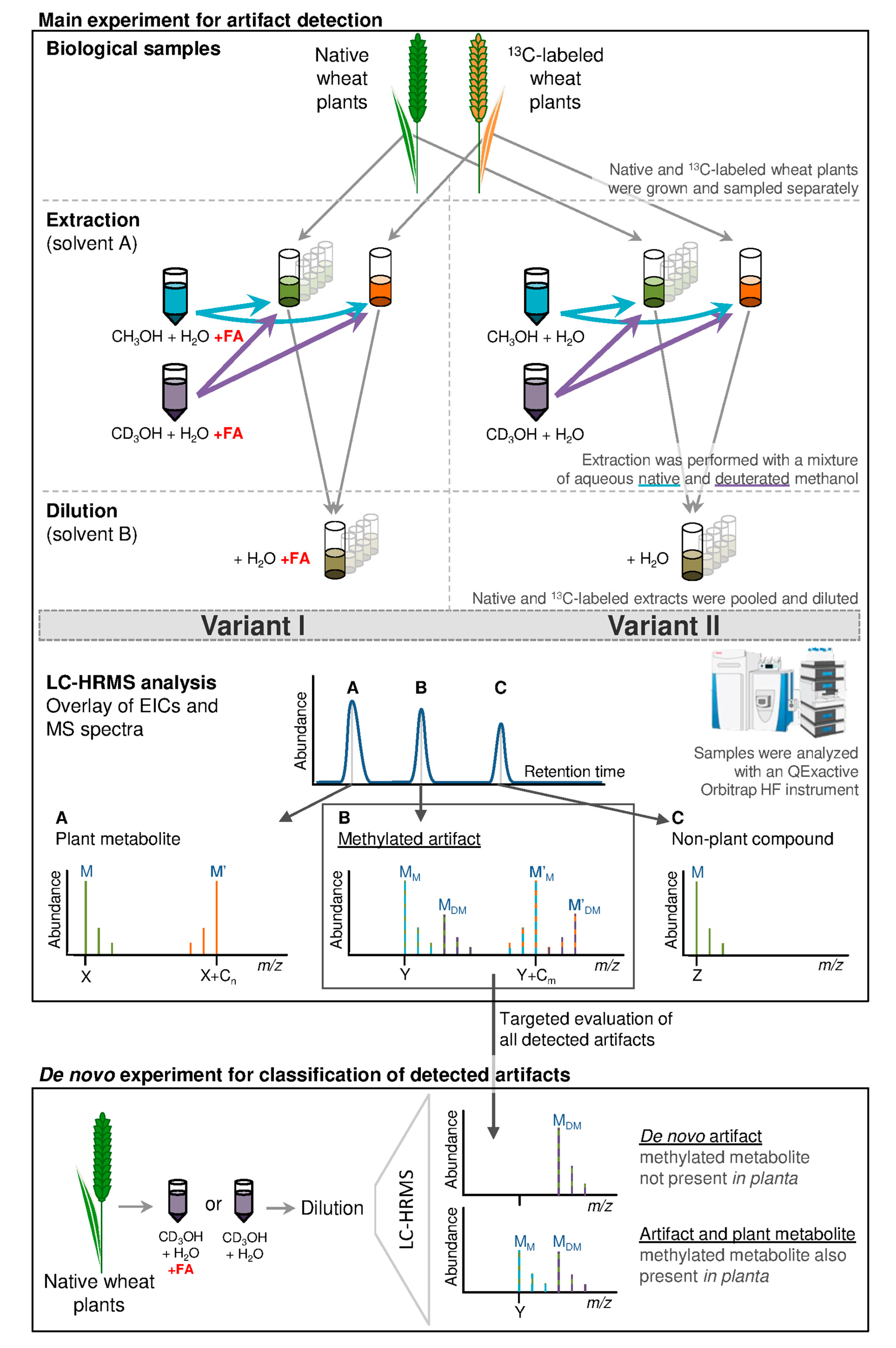
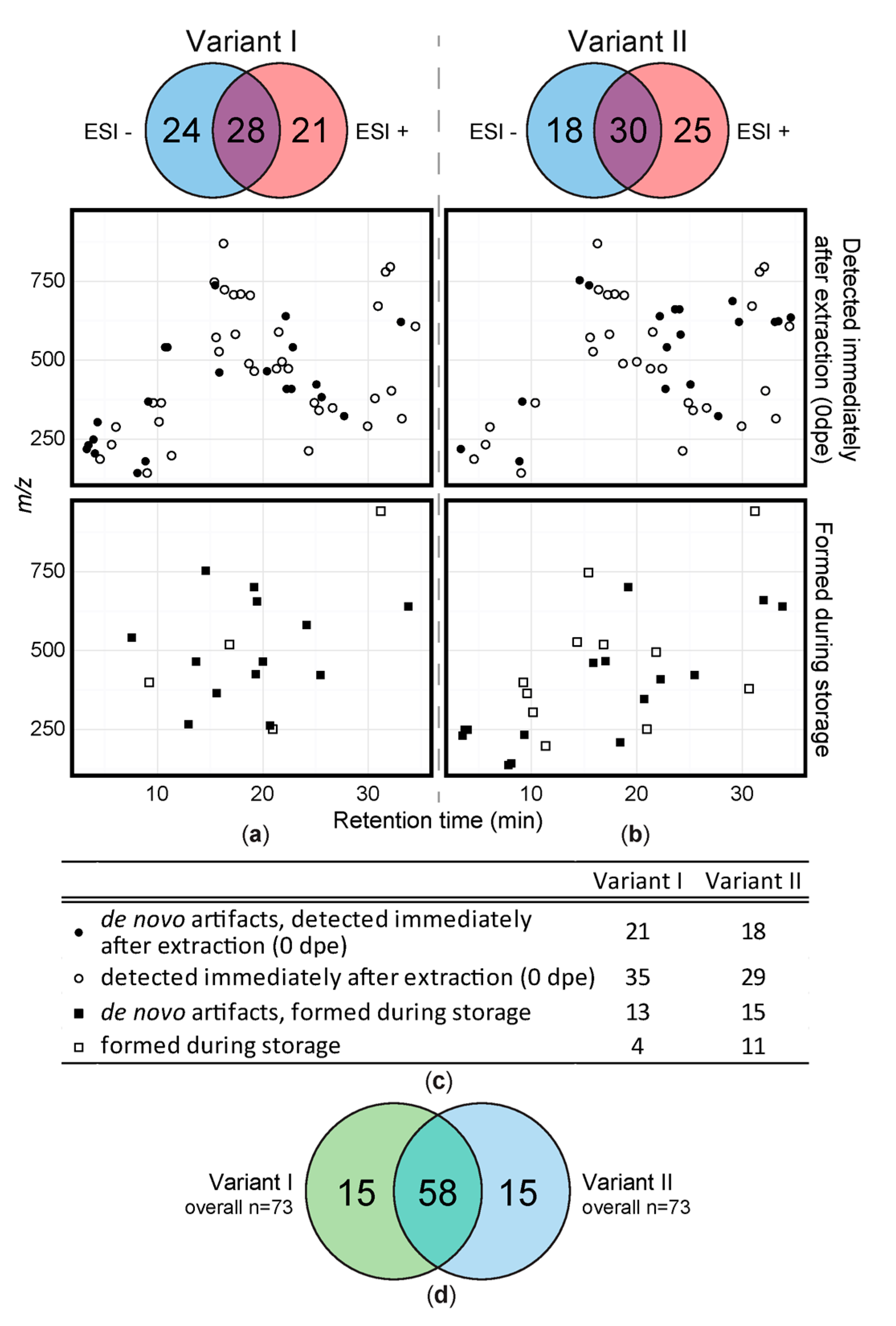
2.1. Data Structure and Overview of Results
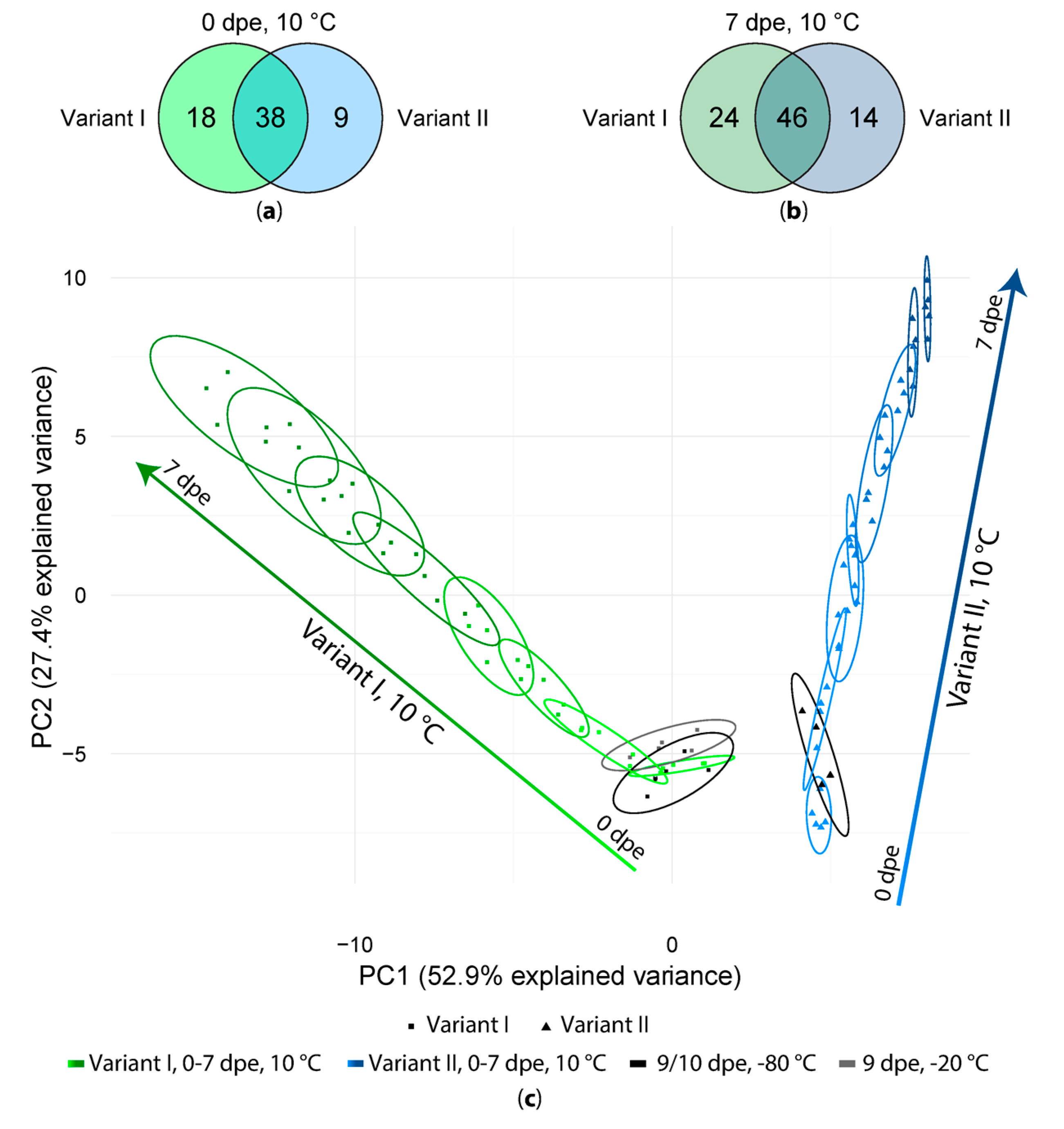
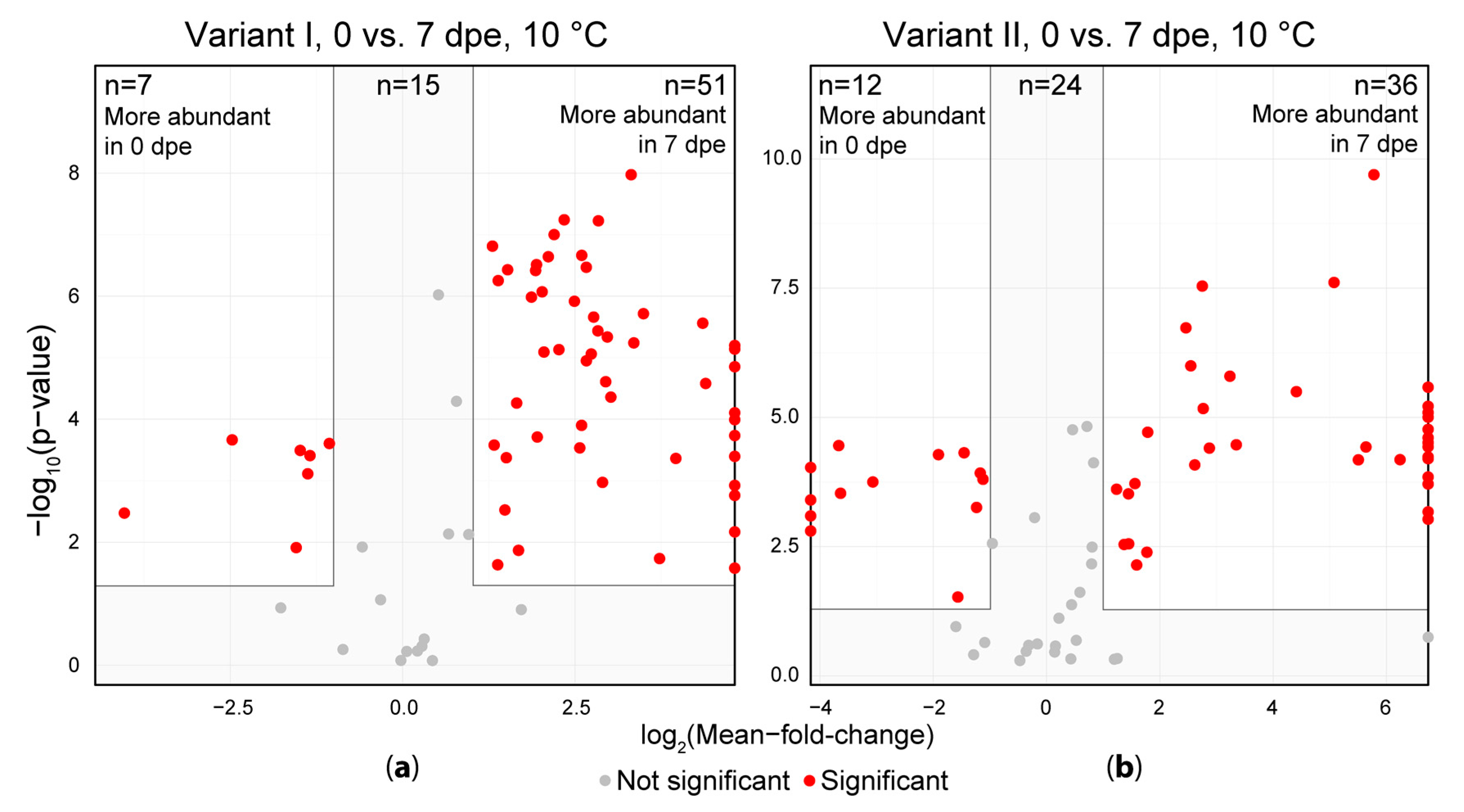
2.2. Short Term Storage of Sample Extracts
2.3. Intensities of Solvent Artifacts
3. Discussion
3.1. Discussion of General Results
3.2. Short Term Storage of Sample Extracts
3.3. Intensities of Solvent Artifacts
4. Materials and Methods
4.1. Chemicals
4.2. Cultivation and Sampling of Plant Material
4.3. Sample Preparation and Extraction of Plant Material
4.4. Preparation of Methylated Citric Acid and Cis-Aconitic Acid
4.5. Influence of Storage Time and Conditions
4.6. LC-HRMS Analysis of Wheat Leaf Samples
4.7. Data Processing
4.8. Statistical Data Evaluation
4.8.1. Selection of De novo Artifact Compounds
4.8.2. Venn Diagrams
4.8.3. Feature Map
4.8.4. Univariate Comparison between Metabolite Abundances of Two Variants
4.8.5. Abundance Histograms
4.8.6. Principal Component Analysis (PCA)
5. Conclusions
Supplementary Materials
Acknowledgments
Author Contributions
Conflicts of Interest
References
- Mushtaq, M.Y.; Choi, Y.H.; Verpoorte, R.; Wilson, E.G. Extraction for metabolomics: Access to the metabolome. Phytochem. Anal. 2014, 25, 291–306. [Google Scholar] [CrossRef] [PubMed]
- DeHaven, C.D.; Evans, A.M.; Dai, H.; Lawton, K.A. Software techniques for enabling high-throughput analysis of metabolomic datasets. In Metabolomics; Roessner, U., Ed.; InTech: Rijeka, Croatia, 2012; pp. 167–192. [Google Scholar]
- Villas-Boas, S.G.; Nielsen, J.; Smedsgaard, J.; Hansen, M.A.E.; Roessner-Tunali, U. Metabolome Analysis: An Introduction; Wiley: Hoboken, NJ, USA, 2007; p. 312. [Google Scholar]
- Doppler, M.; Kluger, B.; Bueschl, C.; Schneider, C.; Krska, R.; Delcambre, S.; Hiller, K.; Lemmens, M.; Schuhmacher, R. Stable isotope-assisted evaluation of different extraction solvents for untargeted metabolomics of plants. Int. J. Mol. Sci. 2016, 17. [Google Scholar] [CrossRef] [PubMed]
- Maltese, F.; van der Kooy, F.; Verpoorte, R. Solvent derived artifacts in natural products chemistry. Nat. Prod. Commun. 2009, 4, 447–454. [Google Scholar] [PubMed]
- Gu, J.-Q.; Li, W.; Kang, Y.-H.; Su, B.-N.; Fong, H.H.S.; van Breemen, R.B.; Pezzuto, J.M.; Kinghorn, A.D. Minor withanolides from Physalis philadelphica: Structures, quinone reductase induction activities and liquid chromatography (lc)-ms-ms investigation as artifacts. Chem. Pharm. Bull. 2003, 51, 530–539. [Google Scholar] [CrossRef] [PubMed]
- Verpoorte, R.; Schripsema, J. Isolation, identification and structure elucidation of alkaloids a general overview. In Alkaloids; Linskens, H.F., Jackson, J.F., Eds.; Springer: Berlin/Heidelberg, Germany, 1994; pp. 1–24. [Google Scholar]
- Brondz, I.; Ekeberg, D.; Høiland, K.; Bell, D.S.; Annino, A.R. The real nature of the indole alkaloids in Cortinarius infractus: Evaluation of artifact formation through solvent extraction method development. J. Chromatogr. A 2007, 1148, 1–7. [Google Scholar] [CrossRef] [PubMed]
- Perry, N.B.; Burgess, E.J.; Foster, L.M.; Gerard, P.J.; Toyota, M.; Asakawa, Y. Insect antifeedant sesquiterpene acetals from the liverwort lepidolaena clavigera. 2. Structures, artifacts and activity. J. Nat. Prod. 2008, 71, 258–261. [Google Scholar] [CrossRef] [PubMed]
- Rajniak, J.; Barco, B.; Clay, N.K.; Sattely, E.S. A new cyanogenic metabolite in arabidopsis required for inducible pathogen defence. Nature 2015, 525, 376–379. [Google Scholar] [CrossRef] [PubMed]
- Stemmler, E.A.; Barton, E.E.; Esonu, O.K.; Polasky, D.A.; Onderko, L.L.; Bergeron, A.B.; Christie, A.E.; Dickinson, P.S. C-terminal methylation of truncated neuropeptides: An enzyme-assisted extraction artifact involving methanol. Peptides 2013, 46, 108–125. [Google Scholar] [CrossRef] [PubMed]
- Poulton, J.E. 22—Transmethylation and demethylation reactions in the metabolism of secondary plant products. In Secondary Plant Products; Conn, E.E., Stumpf, P.K., Eds.; Academic Press: San Diego, CA, USA, 1981; Volume 7, pp. 667–723. [Google Scholar]
- Bueschl, C.; Kluger, B.; Lemmens, M.; Adam, G.; Wiesenberger, G.; Maschietto, V.; Marocco, A.; Strauss, J.; Bödi, S.; Thallinger, G.G.; et al. A novel stable isotope labelling assisted workflow for improved untargeted lc–hrms based metabolomics research. Metabolomics 2014, 10, 754–769. [Google Scholar] [CrossRef] [PubMed]
- Stupp, G.S.; Clendinen, C.S.; Ajredini, R.; Szewc, M.A.; Garret, T.; Menger, R.F.; Yost, R.A.; Beecher, C.; Edison, A.S. Isotopic ratio outlier analysis global metabolomics of caenorhabditis elegans. Anal. Chem. 2013, 85, 11858–11865. [Google Scholar] [CrossRef] [PubMed]
- Giavalisco, P.; Li, Y.; Matthes, A.; Eckhardt, A.; Hubberten, H.; Hesse, H.; Segu, S.; Hummel, J.; Köhl, K.; Willmitzer, L. Elemental formula annotation of polar and lipophilic metabolites using 13C, 15N and 34S isotope labelling, in combination with high-resolution mass spectrometry. Plant J. 2011, 68, 364–376. [Google Scholar] [CrossRef] [PubMed]
- Hegeman, A.D. Plant metabolomics-meeting the analytical challenges of comprehensive metabolite analysis. Brief. Funct. Genom. 2010, 9, 139–148. [Google Scholar] [CrossRef] [PubMed]
- Mahieu, N.G.; Patti, G.J. Systems-level annotation of a metabolomics data set reduces 25000 features to fewer than 1000 unique metabolites. Anal. Chem. 2017, 89, 10397–10406. [Google Scholar] [CrossRef] [PubMed]
- Bueschl, C.; Kluger, B.; Neumann, N.K.N.; Doppler, M.; Maschietto, V.; Thallinger, G.G.; Meng-Reiterer, J.; Krska, R.; Schuhmacher, R. Metextract ii: A software suite for stable isotope assisted untargeted metabolomics. Anal. Chem. 2017, in press. [Google Scholar] [CrossRef] [PubMed]
- R Development Core Team. R: A Language and Environment for Statistical Computing; R Foundation for Statistical Computing: Vienna, Austria, 2008. [Google Scholar]
- Eilertsen, H.C.; Huseby, S.; Degerlund, M.; Eriksen, G.K.; Ingebrigtsen, R.A.; Hansen, E. The effect of freeze/thaw cycles on reproducibility of metabolic profiling of marine microalgal extracts using direct infusion high-resolution mass spectrometry (hr-ms). Molecules 2014, 19, 16373–16380. [Google Scholar] [CrossRef] [PubMed]
- Pinto, J.; Domingues, M.R.; Galhano, E.; Pita, C.; Almeida Mdo, C.; Carreira, I.M.; Gil, A.M. Human plasma stability during handling and storage: Impact on nmr metabolomics. Analyst 2014, 139, 1168–1177. [Google Scholar] [CrossRef] [PubMed]
- Hoagland, D.R.; Arnon, D.I. The water-culture method for growing plants without soil. Circ. Calif. Agric. Exp. Station 1950, 347, 1–32. [Google Scholar]
- Kessner, D.; Chambers, M.; Burke, R.; Agus, D.; Mallick, P. Proteowizard: Open source software for rapid proteomics tools development. Bioinformatics 2008, 24, 2534–2536. [Google Scholar] [CrossRef] [PubMed]
- Pedrioli, P.G.; Eng, J.K.; Hubley, R.; Vogelzang, M.; Deutsch, E.W.; Raught, B.; Pratt, B.; Nilsson, E.; Angeletti, R.H.; Apweiler, R.; et al. A common open representation of mass spectrometry data and its application to proteomics research. Nat. Biotechnol. 2004, 22, 1459–1466. [Google Scholar] [CrossRef] [PubMed]
- Van den Berg, R.A.; Hoefsloot, H.C.; Westerhuis, J.A.; Smilde, A.K.; van der Werf, M.J. Centering, scaling and transformations: Improving the biological information content of metabolomics data. BMC Genom. 2006, 7, 142. [Google Scholar] [CrossRef] [PubMed]
- Wehrens, R. Chemometrics with R: Multivariate Data Analysis in the Natural Sciences and Life Sciences; Springer: Berlin/Heidelberg, Germany, 2011; p. 286. [Google Scholar]
- Murdoch, D.J.; Chow, E.D. A graphical display of large correlation matrices. Am. Stat. 1996, 50, 178–180. [Google Scholar]
| ID | Methylated Form = Artifact | Variant I (+FA) | Variant II (−FA) | |||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| −20 °C | −80 °C | 10 °C | −80 °C | 10 °C | ||||||||||||||||||||||||||||||||||
| Storage Time [dpe] (Days Past Extraction) | ||||||||||||||||||||||||||||||||||||||
| m/z of MM | Rt [min] | De novo | 9 | 9 | 0 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 10 | 0 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | |||||||||||||||||
| 1 | [+]: 218.9587 | 3.30 | x | |||||||||||||||||||||||||||||||||||
| 2 | [−]: 231.0509 | 3.45 | x | |||||||||||||||||||||||||||||||||||
| 3 | [+]: 248.9693 | 3.65 | x | |||||||||||||||||||||||||||||||||||
| 4 | [−]: 249.0614 | 3.93 | x | |||||||||||||||||||||||||||||||||||
| 5 ** | [−]: 205.0353 | 4.07 | x | |||||||||||||||||||||||||||||||||||
| 6 | [−]: 303.9520 | 4.32 | x | |||||||||||||||||||||||||||||||||||
| 7 ** | [−]: 187.0246 | 4.55 | × | |||||||||||||||||||||||||||||||||||
| 8_B | [+]: 232.9745 | 5.65 | x | |||||||||||||||||||||||||||||||||||
| 9_A * | [−]: 143.0349 | 6.05 | ||||||||||||||||||||||||||||||||||||
| 10 | [−]: 541.1667 | 7.58 | ||||||||||||||||||||||||||||||||||||
| 11 * | [+]: 138.0550 | 7.81 | ||||||||||||||||||||||||||||||||||||
| 12_A* | [−]: 143.0349 | 8.10 | ||||||||||||||||||||||||||||||||||||
| 13 | [+]: 180.1019 | 8.86 | ||||||||||||||||||||||||||||||||||||
| 14 | [−]: 143.0348 | 9.02 | ||||||||||||||||||||||||||||||||||||
| 15_C | [M + H]+: 369.1653 | 9.13 | ||||||||||||||||||||||||||||||||||||
| 16 | [+]: 399.1758 | 9.23 | ||||||||||||||||||||||||||||||||||||
| 17 * | [+]: 234.1338 | 9.33 | ||||||||||||||||||||||||||||||||||||
| 18_A | [M − H]−: 365.1352 | 9.59 | x | |||||||||||||||||||||||||||||||||||
| 19_B | [M + H]+: 305.1605 | 10.15 | ||||||||||||||||||||||||||||||||||||
| 20_A | [M − H]−: 365.1351 | 10.36 | x | |||||||||||||||||||||||||||||||||||
| 21 | [−]: 541.1668 | 10.73 | x | |||||||||||||||||||||||||||||||||||
| 22 | [−]: 541.1666 | 10.95 | x | |||||||||||||||||||||||||||||||||||
| 23 * | [+]: 198.1124 | 11.34 | x | |||||||||||||||||||||||||||||||||||
| 24 | [+]: 267.1203 | 12.93 | x | |||||||||||||||||||||||||||||||||||
| 25 | [−]: 465.2332 | 13.67 | x | |||||||||||||||||||||||||||||||||||
| 26 | [−]: 527.1873 | 14.34 | ||||||||||||||||||||||||||||||||||||
| 27 | [−]: 753.2617 | 14.57 | x | |||||||||||||||||||||||||||||||||||
| 28 | [+]: 747.2841 | 15.40 | x | |||||||||||||||||||||||||||||||||||
| 29_A | [M − H]−: 737.3043 | 15.47 | x | |||||||||||||||||||||||||||||||||||
| 30 | [−]: 572.2254 | 15.56 | ||||||||||||||||||||||||||||||||||||
| 31 | [−]: 365.1351 | 15.62 | x | |||||||||||||||||||||||||||||||||||
| 32 | [−]: 527.1877 | 15.84 | ||||||||||||||||||||||||||||||||||||
| 33_A | [−]: 461.2386 | 15.87 | x | |||||||||||||||||||||||||||||||||||
| 34 | [−]: 869.3465 | 16.26 | x | |||||||||||||||||||||||||||||||||||
| 35_B | [M − H]−: 723.2886 | 16.36 | ||||||||||||||||||||||||||||||||||||
| 36 | [+]: 519.2415 | 16.83 | x | |||||||||||||||||||||||||||||||||||
| 37 | [−]: 467.1012 | 17.03 | x | |||||||||||||||||||||||||||||||||||
| 38 | [−]: 707.2937 | 17.23 | ||||||||||||||||||||||||||||||||||||
| 39_C | [M − H]−: 737.2678 | 17.40 | x | |||||||||||||||||||||||||||||||||||
| 40_D | [M + H]+: 709.3071 | 17.90 | x | |||||||||||||||||||||||||||||||||||
| 41 * | [+]: 209.0811 | 18.40 | x | |||||||||||||||||||||||||||||||||||
| 42_C | [−]: 489.2337 | 18.68 | ||||||||||||||||||||||||||||||||||||
| 43_B | [M − H]−: 705.2776 | 18.80 | x | |||||||||||||||||||||||||||||||||||
| 44 | [−]: 701.2082 | 19.16 | x | |||||||||||||||||||||||||||||||||||
| 45 | [−]: 465.1795 | 19.18 | ||||||||||||||||||||||||||||||||||||
| 46 | [+]: 425.2140 | 19.34 | x | |||||||||||||||||||||||||||||||||||
| 47 | [−]: 655.3539 | 19.45 | ||||||||||||||||||||||||||||||||||||
| 48 | [+]: 495.2195 | 19.99 | x | |||||||||||||||||||||||||||||||||||
| 49 | [−]: 465.1793 | 20.01 | x | |||||||||||||||||||||||||||||||||||
| 50 | [−]: 465.1798 | 20.39 | ||||||||||||||||||||||||||||||||||||
| 51 | [+]: 263.1616 | 20.66 | x | |||||||||||||||||||||||||||||||||||
| 52 | [−]: 347.1244 | 20.69 | ||||||||||||||||||||||||||||||||||||
| 53_B | [+]: 251.1253 | 20.94 | x | |||||||||||||||||||||||||||||||||||
| 54_D | [−]: 473.2390 | 21.29 | x | |||||||||||||||||||||||||||||||||||
| 55 | [−]: 589.2859 | 21.50 | x | |||||||||||||||||||||||||||||||||||
| 56_C | [M + H]+, [M + Na]+: 495.2196 | 21.81 | x | |||||||||||||||||||||||||||||||||||
| 57 | [−]: 639.359 | 22.17 | ||||||||||||||||||||||||||||||||||||
| 58_A | [M + Na]+: 409.2193 | 22.25 | ||||||||||||||||||||||||||||||||||||
| 59_B | [−]: 473.2390 | 22.41 | ||||||||||||||||||||||||||||||||||||
| 60 | [+]: 409.2193 | 22.71 | ||||||||||||||||||||||||||||||||||||
| 61_D * | [M + H]+: 541.1699 | 22.84 | ||||||||||||||||||||||||||||||||||||
| 62 | [+]: 661.2500 | 23.62 | ||||||||||||||||||||||||||||||||||||
| 63 | [+]: 661.2500 | 24.03 | x | |||||||||||||||||||||||||||||||||||
| 64_A * | [M − H]−: 581.1662 | 24.14 | ||||||||||||||||||||||||||||||||||||
| 65_B * | [M + Na]+: 235.1303 | 24.35 | ||||||||||||||||||||||||||||||||||||
| 66_C | [M + Na]+: 365.2296 | 24.88 | ||||||||||||||||||||||||||||||||||||
| 67 | [+]: 423.2348 | 25.08 | ||||||||||||||||||||||||||||||||||||
| 68_A | [M − H]−: 341.233 | 25.32 | ||||||||||||||||||||||||||||||||||||
| 69 | [+]: 423.2349 | 25.48 | ||||||||||||||||||||||||||||||||||||
| 70 | [+]: 383.2193 | 25.57 | ||||||||||||||||||||||||||||||||||||
| 71_B | [M + Na]+: 349.1982 | 26.61 | ||||||||||||||||||||||||||||||||||||
| 72 | [−]: 323.2227 | 27.71 | ||||||||||||||||||||||||||||||||||||
| 73_B | [−]: 687.2312 | 29.07 | x | |||||||||||||||||||||||||||||||||||
| 74 | [−]: 621.2721 | 29.68 | x | |||||||||||||||||||||||||||||||||||
| 75_B | [+]: 331.224 | 29.93 | x | |||||||||||||||||||||||||||||||||||
| 76 | [+]: 379.2839 | 30.63 | ||||||||||||||||||||||||||||||||||||
| 77 | [+]: 671.4276 | 30.92 | x | |||||||||||||||||||||||||||||||||||
| 78 | [+]: 941.5072 | 31.18 | ||||||||||||||||||||||||||||||||||||
| 79_A | [+]: 779.4543 | 31.64 | x | |||||||||||||||||||||||||||||||||||
| 80 | [+]: 659.2468 | 31.99 | ||||||||||||||||||||||||||||||||||||
| 81 | [+]: 403.2817 | 32.19 | x | |||||||||||||||||||||||||||||||||||
| 82_C | [M + Na]+, [M + CH3OH + H]+: 795.4858 | 32.08 | x | |||||||||||||||||||||||||||||||||||
| 83_B * | [M + Na]+: 315.2293 | 33.18 | x | |||||||||||||||||||||||||||||||||||
| 84_B * | [M + H]+: 621.2703 | 33.09 | x | |||||||||||||||||||||||||||||||||||
| 85_B | [M + H]+: 623.2858 | 33.41 | x | |||||||||||||||||||||||||||||||||||
| 86 * | [+]: 639.2806 | 33.79 | x | |||||||||||||||||||||||||||||||||||
| 87_B | [M + H]+: 607.2911 | 34.47 | x | |||||||||||||||||||||||||||||||||||
| 88 | [−]: 635.2514 | 34.59 | ||||||||||||||||||||||||||||||||||||
| Color code: | not present | <1×103 | 1×103–1×104 | 1×104–1×105 | 1×105–1×106 | 1×106–1×107 | 1×107–1×108 | |||||||||||||||||||||||||||||||
© 2017 by the authors. Licensee MDPI, Basel, Switzerland. This article is an open access article distributed under the terms and conditions of the Creative Commons Attribution (CC BY) license (http://creativecommons.org/licenses/by/4.0/).
Share and Cite
Sauerschnig, C.; Doppler, M.; Bueschl, C.; Schuhmacher, R. Methanol Generates Numerous Artifacts during Sample Extraction and Storage of Extracts in Metabolomics Research. Metabolites 2018, 8, 1. https://doi.org/10.3390/metabo8010001
Sauerschnig C, Doppler M, Bueschl C, Schuhmacher R. Methanol Generates Numerous Artifacts during Sample Extraction and Storage of Extracts in Metabolomics Research. Metabolites. 2018; 8(1):1. https://doi.org/10.3390/metabo8010001
Chicago/Turabian StyleSauerschnig, Claudia, Maria Doppler, Christoph Bueschl, and Rainer Schuhmacher. 2018. "Methanol Generates Numerous Artifacts during Sample Extraction and Storage of Extracts in Metabolomics Research" Metabolites 8, no. 1: 1. https://doi.org/10.3390/metabo8010001
APA StyleSauerschnig, C., Doppler, M., Bueschl, C., & Schuhmacher, R. (2018). Methanol Generates Numerous Artifacts during Sample Extraction and Storage of Extracts in Metabolomics Research. Metabolites, 8(1), 1. https://doi.org/10.3390/metabo8010001